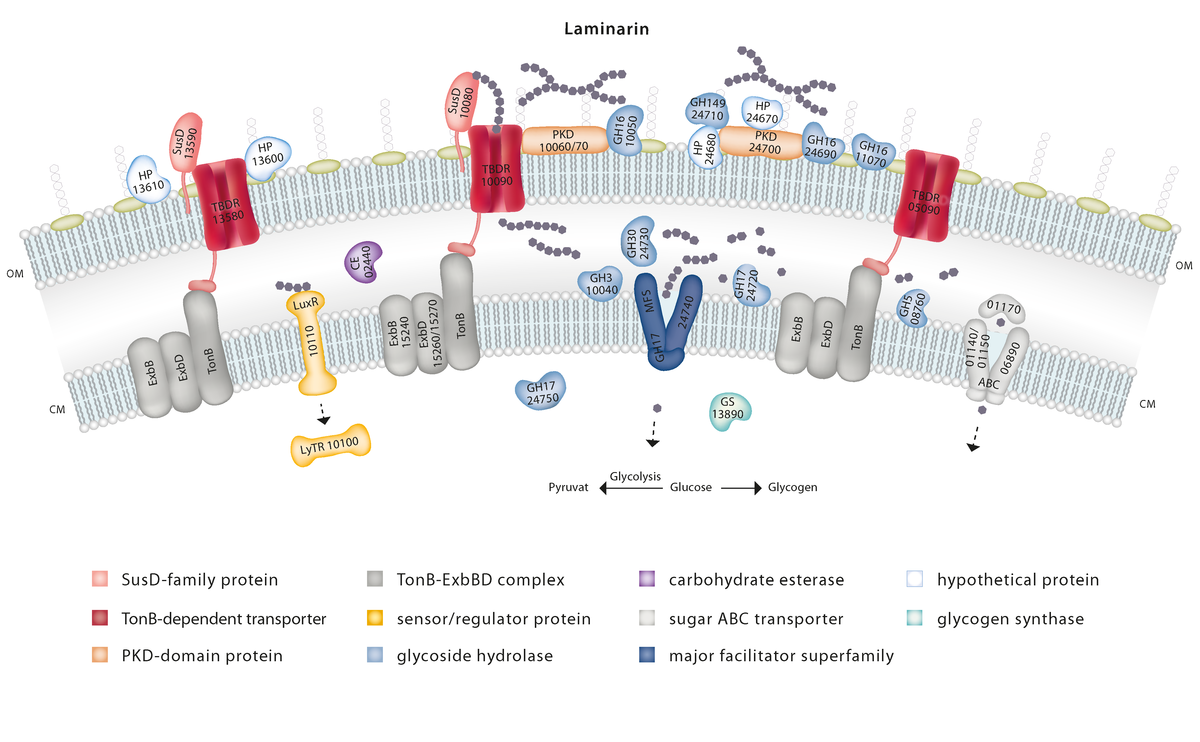
Molekulare Mechanismen des marinen Polysaccharidabbaus
Algenblüten, d. h. eine massive Vermehrung von Algen in marinen Ökosystemen, werden durch jahreszeitlich bedingte Veränderungen der Umweltbedingungen und durch Nährstoffeinträge der extensiven Landwirtschaft verursacht. Große Mengen an Algenbiomasse wirken sich nachteilig auf die Küstenökosysteme, den Tourismus aber auch Aquakulturen aus. Gleichzeitig stellen sie eine bislang noch unzureichend genutzte, nachhaltige Ressource für seltene marine Zucker dar. Das komplexe Spektrum an Nährstoffen, das von Algen freigesetzt wird, bietet Bakterien einen fruchtbaren Nährboden. Diese Nährstoffe umfassen strukturell vielseitige Algenpolysaccharide. Um diese teilweise widerspenstige Nahrungsquelle zu erschließen, verfügen Bakteriengemeinschaften über ein vielfältiges Repertoire an hochspezialisierten Enzymen, substratbindenden Proteinen und Transportern. Interessanterweise verfolgen einzelne Bakterien ganz unterschiedliche Strategien, um diese Algeninhaltsstoffe zu verwerten, sich gegen Nahrungskonkurrenten durchzusetzen oder voneinander zu profitieren. Mit diesen bakteriellen Strategien beschäftigt sich unsere DFG-Forschungsgruppe POMPU (FOR 2406; www.pompu-project.de).
In Zusammenarbeit mit dem Max-Planck-Institut für Marine Mikrobiologie (Bremen) und dem Alfred-Wegener-Institut (AWI), Helmholtz-Zentrum für Polar- und Meeresforschung (Bremerhaven) untersuchen wir Mikroalgenblüten am Langzeitbeprobungsort Kabeltonne (Helgoland Roads) in der Nähe der Insel Helgoland in der Deutschen Bucht (Nordsee). Eine massive Vermehrung von Mikroalgen in der Wassersäule wird von einem dramatischen Anstieg der Bakterienbiomasse begleitet. Bei näherer Betrachtung der Bakteriengemeinschaft beobachteten wir einerseits eine spezifische Abfolge einzelner Bakteriengruppen und andererseits das Wiederauftreten einzelner Stämme innerhalb dieses Musters über mehrere Jahre der Probenahme. Unter den Bakteriengruppen, die die Algenblüte dominierten, befanden sich Vertreter der Flavobakterien, die in der Lage sind, eine Vielzahl von Algenpolysacchariden abzubauen. Im Verlauf einer Algenblüte nutzen Bakterien vorzugsweise zunächst leicht verdauliche Kohlenhydrate, bevor sie strukturell komplexere Algenpolysaccharide verwerten. Die entsprechenden Gene, die die Proteine für den Abbau der Polysaccharide codieren, sind dabei häufig im Genom in sogenannten "polysaccharide-utilization loci" (PULs) zu finden. Da nicht jedes Bakterium das gesamte Spektrum an Polysacchariden nutzen kann, braucht es verschiedene Bakteriengruppen mit individuellen Substratspezifitäten, um letztlich die gesamte Algenbiomasse in einer Art Mannschaftsleistung umzusetzen. Dieses Konsortium vereint Bakterien mit verschiedenen Wachstumsstrategien, d. h. sowohl Generalisten mit vielen PULs für den Abbau verschiedener Substrate als auch Spezialisten mit wenigen spezifischen PULs. Jede Strategie erfordert individuelle Anpassungen, die es zu erforschen gilt.
Um bakterielle Mechanismen und mikrobielle Interaktionen innerhalb einer Algenblüte zu verstehen, sind Metagenom- und Metaproteomanalysen zwei essentielle methodische Säulen des Forschungsschwerpunktes. Ein weiterer wichtiger Aspekt ist die genaue Untersuchung von Schlüsselbakterien, die in Algenblüten isoliert wurden. Wir sind vor allem an der funktionellen Charakterisierung von PULs und Abbauenzymen dieser Polysaccharid-verwertenden Bakterien interessiert. Um die molekularen Mechanismen des Polysaccharidabbaus zu entschlüsseln nutzen wir u.a. Proteomanalysen sowie verschiedene molekularbiologische, analytische und mikroskopische Methoden. So können wir Enzyme identifizieren, die die gezielte Zerlegung von Polysacchariden in definierte Oligo- und Monomere katalysieren. Diese Enzyme werden auf potenzielle pharmazeutische und biotechnologische Anwendungen getestet. Diese Arbeiten werden in enger Kooperation mit den Instituten für Mikrobiologie sowie Biochemie der Universität Greifswald und der Station Biologique de Roscoff in Frankreich durchgeführt.
Verantwortliche Mitarbeiter:
Dr. Marie-Katherin Zühlke, Dr. Alexandra Dürwald, Irena Beidler, Daniel Bartosik
Literatur:
- Kappelmann L, Krüger K, Hehemann JH, Harder J, Markert S, Unfried F, Becher D, Shapiro N, Schweder T, Amann RI, Teeling H. 2019. Polysaccharide utilization loci of North Sea Flavobacteriia as basis for using SusC/D-protein expression for predicting major phytoplankton glycans. ISME J. 13(1):76-91.
- Koch H, Dürwald A, Schweder T, Noriega-Ortega B, Vidal-Melgosa S, Hehemann JH, Dittmar T, Freese HM, Becher D, Simon M, Wietz M. 2019. Biphasic cellular adaptations and ecological implications of Alteromonas macleodii degrading a mixture of algal polysaccharides. ISME J. 13(1):92-103.
- Unfried F, Becker S, Robb CS, Hehemann JH, Markert S, Heiden SE, Hinzke T, Becher D, Reintjes G, Krüger K, Avcı B, Kappelmann L, Hahnke RL, Fischer T, Harder J, Teeling H, Fuchs B, Barbeyron T, Amann RI, Schweder T. 2018. Adaptive mechanisms that provide competitive advantages to marine bacteroidetes during microalgal blooms. ISME J. 12(12):2894-2906.
- Chen J, Robb CS, Unfried F, Kappelmann L, Markert S, Song T, Harder J, Avcı B, Becher D, Xie P, Amann RI, Hehemann JH, Schweder T, Teeling H. 2018. Alpha- and beta-mannan utilization by marine Bacteroidetes. Environ Microbiol. 20(11):4127-4140.
- Reisky L, Büchsenschütz HC, Engel J, Song T, Schweder T, Hehemann JH, Bornscheuer UT. 2018. Oxidative demethylation of algal carbohydrates by cytochrome P450 monooxygenases. Nat Chem Biol. 14(4):342-344.
- Hehemann JH, Truong LV, Unfried F, Welsch N, Kabisch J, Heiden SE, Junker S, Becher D, Thürmer A, Daniel R, Amann R, Schweder T. 2017. Aquatic adaptation of a laterally acquired pectin degradation pathway in marine Gammaproteobacteria. Environ Microbiol. 19(6):2320-2333.
- Kabisch A, Otto A, König S, Becher D, Albrecht D, Schüler M, Teeling H, Amann RI, Schweder T. 2014. Functional characterization of polysaccharide utilization loci in the marine Bacteroidetes 'Gramella forsetii' KT0803. ISME J. 8(7):1492-502.
- Teeling H, Fuchs BM, Becher D, Klockow C, Gardebrecht A, Bennke CM, Kassabgy M, Huang S, Mann AJ, Waldmann J, Weber M, Klindworth A, Otto A, Lange J, Bernhardt J, Reinsch C, Hecker M, Peplies J, Bockelmann FD, Callies U, Gerdts G, Wichels A, Wiltshire KH, Glöckner FO, Schweder T, Amann R. 2012. Substrate-Controlled Succession of Marine Bacterioplankton Populations Induced by a Phytoplankton Bloom. Science. 336(6081):608-11.