Funktionelle Analyse und Stammoptimierung industriell relevanter Bacilli
Bacillus subtilis gehört zu den am besten untersuchten Bakterien und ist vor allem für die Fähigkeit bekannt, unter extremen Umweltbedingungen metabolisch inaktive Sporen als Überdauerungsform zu bilden. Zusätzlich ist B. subtilis einMeister in der Ausbildung weiterer differenzierter Zelltypen, welche bei der Beweglichkeit, der Biofilmbildung oder der Aufnahme freier DNA aus der Umgebung eine wichtige Rolle spielen. Neben dieser Zelldifferenzierung sind Bacilli in der Lage, große Mengen biotechnologisch relevanter Substanzen zu sekretieren, weshalb Vertreter wie z.B. B. subtilis oder B. licheniformis für die industrielle Produktion von Vitaminen, Enzymen oder Sekundärmetaboliten unverzichtbar geworden sind.
In enger Zusammenarbeit mit unseren akademischen und industriellen Partnern analysieren wir die Physiologie diverser Bacilli und modifizieren diese Zellfabriken. Dabei erhalten wir mithilfe unterschiedlicher Methoden Einblicke in das Wachstums- und Produktionsverhalten, die Expression und Regulation ausgewählter Gene oder visualisieren spezifische Differenzierungsprozesse einzelner Zellen. Molekulare Techniken zur Klonierung und Genomeditierung (z.B. CRISPR/Cas oder Cre/lox) ermöglichen es uns zudem neue Stammvarianten zu erstellen und bestehende Bacillus-Expressionsplattformen zu optimieren.
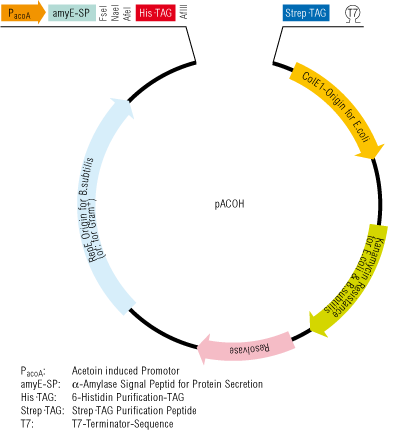
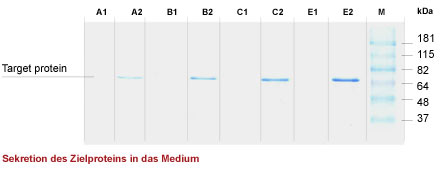
Abb. 1. Schematische Darstellung eines Acetoin-regulierten Bacillus subtilis-Expressionssystems sowie ein Beispiel der extrazellulären Proteinproduktion mit diesem System
Verantwortliche Mitarbeiter:
Dr. Britta Jürgen, Dr. Norma Welsch, Maximilian Hilkmann, Jana Matulla
Literatur:
- Trung NT, Hung NM, Thuan NH, Canh NX, Schweder T, Jürgen B. 2019. An auto-inducible phosphate-controlled expression system of Bacillus licheniformis. BMC Biotechnol. 19(1):3.
- Handtke S, Albrecht D, Zühlke D, Otto A, Becher D, Schweder T, Riedel K, Hecker M, Voigt B. 2017. Bacillus pumilus KatX2 confers enhanced hydrogen peroxide resistance to a Bacillus subtilis PkatA::katX2 mutant strain. Microb Cell Fact. 16(1):72.
- Kumpfmüller J, Methling K, Fang L, Pfeifer BA, Lalk M, Schweder T. 2015. Production of the polyketide 6-deoxyerythronolide B in the heterologous host Bacillus subtilis. Appl Microbiol Biotechnol. 100(3):1209-20.
- Zobel S, Kumpfmüller J, Süssmuth RD, Schweder T. 2014. Bacillus subtilis as heterologous host for the secretory production of the non-ribosomal cyclodepsipeptide enniatin. Appl Microbiol Biotechnol. 99(2):681-91.
- Voigt B, Schroeter R, Schweder T, Jürgen B, Albrecht D, van Dijl JM, Maurer KH, Hecker M. 2014. A proteomic view of cell physiology of the industrial workhorse Bacillus licheniformis. J Biotechnol. 191:139-49.
- Handtke S, Schroeter R, Jürgen B, Methling K, Schlüter R, Albrecht D, van Hijum SA, Bongaerts J, Maurer KH, Lalk M, Schweder T, Hecker M, Voigt B. 2014. Bacillus pumilus reveals a remarkably high resistance to hydrogen peroxide provoked oxidative stress. PLoS One. 9(1):e85625.
- Kumpfmüller J, Kabisch J, Schweder T. 2013. An optimized technique for rapid genome modifications of Bacillus subtilis. J Microbiol Methods. 95(3):350-2.
- Kabisch J, Pratzka I, Meyer H, Albrecht D, Lalk M, Ehrenreich A, Schweder T. 2013. Metabolic engineering of Bacillus subtilis for growth on overflow metabolites. Microb Cell Fact. 12:72.
- Kabisch J, Thürmer A, Hübel T, Popper L, Daniel R, Schweder T. 2013. Characterization and optimization of Bacillus subtilis ATCC 6051 as an expression host. J Biotechnol. 163(2):97-104.
- Enfors SO, Jahic M, Rozkov A, Xu B, Hecker M, Jürgen B, Krüger E, Schweder T, Hamer G, O'Beirne D, Noisommit-Rizzi N, Reuss M, Boone L, Hewitt C, McFarlane C, Nienow A, Kovacs T, Trägårdh C, Fuchs L, Revstedt J, Friberg PC, Hjertager B, Blomsten G, Skogman H, Hjort S, Hoeks F, Lin HY, Neubauer P, van der Lans R, Luyben K, Vrabel P, Manelius Å. 2001. Physiological responses to mixing in large scale bioreactors. J Biotechnol. 85(2):175-85.
